1. Introdução
Circovírus suíno tipo 2 (PCV2) é o agente etiológico das doenças associadas a circovirose suína (PCVD). O PCV2 foi identificado em 1998 em tecidos de suínos que sofriam de síndrome multissistêmica de ausência de crescimento pós-desmame (PMWS)(1, 2), uma síndrome inicialmente observada em animais saudáveis no Canadá em 1995. Desde então, o PCV2 tem sido associado a diversas manifestações clínicas, incluindo doenças sistêmicas, entéricas, respiratórias e reprodutivas, assim como a dermatite suína e síndrome de nefropatia, que agora são reconhecidas como doença associada ao PCV2, PCVAD ou PCVD(3, 4). PMWS é a manifestação clínica mais prevalente e severa da infecção por PCV2, sendo caracterizada por perdas e lesões graves no tecido linfoide, classificadas como sistêmicas ou subclínicas. Embora a infecção pelo PCV2 tenha sido identificada pela primeira vez no ano 2000(5), estudos retrospectivos demonstraram que o vírus já estava presente desde 1978(6). Desde a sua detecção inicial, a infecção por PCV2 tem sido amplamente distribuída nos rebanhos suínos brasileiros causando perdas sanitárias e de produção(7). Embora a PCVD possa ser controlada através da correção dos fatores de risco e pelo uso de vacinas(4), ela continua sendo um desafio no Brasil, principalmente devido às mudanças na sua apresentação.
O PCV2 pertence ao gênero Circovirus da família Circoviridae (4), e é o menor vírus de DNA de fita simples com replicação autônoma, não envelopada e circular(1). A evolução deste genótipo nos últimos anos tem sido caracterizada por surtos severos de PCVD, relatados na América do Norte de 2004 a 2006. Durante este período, o genoma dos isolados de PCV2 encontrados nos rebanhos afetados foi comparado com os isolados previamente encontrados(8-10). Mais tarde, uma nova variante do PCV2 foi detectada em rebanhos previamente vacinados contra o agente na América do Norte, China, Brasil e em outros países produtores de suínos(11-13). Esta evolução esteve associada a falhas da vacina de PCV2. No Brasil, um mutante de PCV2 associado a falhas vacinais foi identificado pela primeira vez em 2012, e a cepa viral era muito semelhante à encontrada na China entre 2004 e 2008(13). O vírus foi isolado de suínos vacinados contra PCV2 que apresentavam sinais clínicos típicos de PCVD e diagnóstico laboratorial positivo para PCV2. As análises genômicas revelaram adições ou substituições de nucleotídeos resultando em modificações no genoma do vírus, denominado mPCV2. Estas alterações, por sua vez, induziram alterações em aminoácidos localizados nos epítopos responsáveis pela ativação do sistema imunológico. No final da sequência ORF2 (open reading frame 2) para a proteína do capsídeo viral, a variante apresentou nucleotídeos adicionais para três aminoácidos extras(13).
Devido a alta variabilidade genômica observada no PCV2, os isolados foram categorizados em nove genótipos, variando de PCV2a a PCV2i, com base na ORF2 ou sequências completas do genoma(14-16). Atualmente existem inúmeras vacinas comerciais disponíveis, sendo a maioria delas baseadas no genótipo “a” (PCV2a)(4, 17). Porém, com o aumento da diversidade genotípica circulante, estão sendo consideradas vacinas mais eficazes que incorporem atualizações e/ou incluam novos genótipos (17). Além do PCV2, muitas doenças relacionadas ao PCDV envolvem outros agentes (por exemplo, circovírus suíno tipo 3, PCV3), agravando os casos clínicos(8). Assim, a investigação de coinfecções por PCV3 e PCV2, incluindo a genotipagem, é essencial nos casos de PCVD clínica(18, 19).
O Brasil é o quarto maior produtor e exportador de carne suína, sendo considerado importante no cenário internacional(20, 21). Apesar da importância da produção suína no Brasil, com mais de 2 milhões de porcas alojadas e 5 milhões de toneladas de carne suína produzidas em 2022(21), poucos laboratórios realizam análises moleculares, especialmente análises especializadas como a genotipagem viral(22). Isto dificulta aos médicos veterinários a interpretação de casos clínicos, especialmente quando as vacinas são utilizadas para controlar doenças como a PCVD. Considerando os prejuízos causados pela infecção por PCV2 na produção suína a nível mundial e o impacto econômico gerado, bem como a detecção de PCV2 em rebanhos vacinados, os objetivos deste trabalho foram caracterizar genótipos de PCV2 para avaliar a presença de PCV2a PCV2b e PCV2d no campo, por região, fase de produção e material coletado, utilizando um método de diagnóstico rápido, de baixo custo e eficaz. Além disso, a presença de coinfecções entre os genótipos e PCV3 também foi investigada nas amostras analisadas.
2. Material e Métodos
2.1 Amostras analisadas
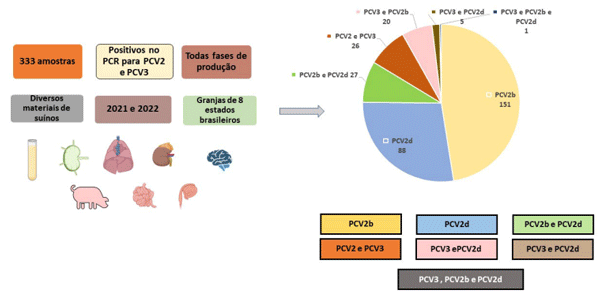
Trezentas e trinta e três amostras de casos clínicos suspeitos de PCVD submetidas ao Centro de Diagnóstico de Sanidade Animal (CEDISA) entre 2021 e 2022, provenientes de oito estados brasileiros (Goiás, Mato Grosso, Mato Grosso do Sul, Minas Gerais, Paraná, Rio Grande do Sul, Santa Catarina e São Paulo) foram utilizados neste estudo. As amostras foram coletadas de diferentes órgãos e materiais de acordo com condições clínicas e compatibilidade com lesões de PCVD. As amostras incluíram 105 amostras de soro, 10 amostras de útero, quatro amostras de esfregaço retal, três amostras de rim, 50 amostras de pulmão, seis amostras de linfonodos, 31 amostras de intestino, 61 amostras de fetos (que incluíam natimortos e fetos mumificados), duas amostras de cérebro e 58 amostras de pool de órgãos (fígado, rim, pulmão, baço, linfonodos e tecido cardíaco). As amostras foram obtidas através de convênio técnico-científico entre o Centro de Pesquisa Embrapa Suínos e Aves e o CEDISA (Convênio 21000.18/0009-7)(22).
Todas as amostras foram submetidas à detecção de DNA viral de PCV2 ou PCV3 através de PCR em tempo real (qPCR). Para isso, as amostras foram pré-tratadas durante a noite com tampão de lise tecidual (ATL, Qiagen, Venlo, Holanda) e proteinase K (Qiagen, Venlo, Holanda) a 56°C sob agitação. O DNA viral foi extraído usando kit IndiMag Pathogen (Indical Bioscience, Leipzig, Alemanha) com um sistema de extração automatizado da IndiMag 48S (Indical Bioscience, Leipzig, Alemanha). O qPCR para diagnóstico de PCV2 foi realizado visando a amplificação de uma região do gene Cap (ORF2) utilizando os primers específicos PCV2_F (5’-CCA GGA GGG GCG TTG TGA CT-3’) e PCV2_R (5’-CGC TAC CGT TGG AGA AGG AA3’) e a sonda PCV2_S (5’-AAT GGC ATC TTC AAC ACC CGC CT CT-3’), como já descrito(23). Para a reação de qPCR, 2,5 µL do DNA extraído foi adicionado ao master mix GoTaq® Probe qPCR (Promega Corporation, Madison, WI, USA), 1,25 µM e 0,5 µM dos primers e sonda, respectivamente. Para PCV3, o gene de replicação conservado foi detectado utilizando os primers PCV3_535_F (5’-TGA CGG AGA CGT CGG GAA AT-3’) e PCV3_465_R (5’-CGG TTT ACC CAA CCC CAT CA-3’), e a sonda PCV3_535_F (5’-TGA CGG AGA CGT CGG GAA AT-3’), conforme descrito em estudo(14). Para a reação, 2 µL do DNA extraído foi adicionado ao master mix GoTaq Probe qPCR (Promega Corporation, Madison, WI, USA), com 0,8 µM e 0,4 µM dos primers específicos e sonda, respectivamente. O sinal de fluorescência foi detectado no final de cada ciclo de extensão no termociclador QuantStudioTM Flex 6 (Applied Biosystems, Foster City, CA, USA). Todas as amostras que apresentaram Ct < 38,00 em ambos os testes foram consideradas positivas.
2.2 Controles positivos de DNA de PCV2 para genotipagem
Para encontrar controles positivos para cada genótipo, 20 amostras foram selecionadas aleatoriamente e submetidas ao sequenciamento. A reação de sequenciamento foi realizada com primers previamente descritos(24), visando amplificar a região ORF2 do PCV2. Após a análise do sequenciamento genômico, foi possível selecionar amostras de controle positivo para os genótipos PCV2b e PCV2d. Para o genótipo PCV2a foi utilizada uma amostra de referência previamente sequenciada da “Coleção de microrganismos de interesse para suínos e aves” (CMISEA) da Embrapa Suínos e Aves(13). As amostras sequenciadas foram testadas para validação dos ensaios de PCR convencional (cPCR). Os resultados dos testes de controle positivo estão ilustrados na Figura 1.
Figura 1 Gel de eletroforese mostrando os produtos de DNA amplificados para os genótipos de PCV2, com seu peso molecular: PCV2a com 277 pb, PCV2b com 277 pb e PCV2d com 343 pb
2.3 PCR convencional (cPCR) utilizado para genotipagem de PCV2
Cada amostra foi testada individualmente para cada um dos três genótipos de interesse do PCV2. A reação de cPCR foi baseada na região ORF2 do PCV2, região que apresenta maiores taxas de diversidade. As sequências dos primers utilizados(25-27) estão descritas na Tabela 1. Os primers foram diluídos para 10 pmol e a PCR utilizou 2,5 µl de tampão, 1 µl com 25 mmol de MgCl2, 1 µl de dNTPs, 0,6 µl de cada primer e 2 µl de DNA de cada amostra. As condições de PCR utilizadas foram as seguintes: desnaturação inicial a 94°C por 1 min, 35 ciclos com desnaturação a 94°C por 30 segundos, anelamento a 60°C por 30 segundos e extensão a 72°C por 1 min, e extensão final a 72°C por 10 min(25-27). Os produtos de cPCR foram submetidos a eletroforese em gel de agarose a 1% e visualizados sob luz UV após coloração com brometo de etídio a 2%. Devido o tamanho idêntico do produto dos genótipos PCV2a e PCV2b, cada amostra foi submetida separadamente em cada reação de cPCR, possibilitando assim a distinção dos genótipos.
Tabela 1 Primers utilizados na análise de genotipagem de PCV2.
2.4 Genotipagem de PCV2 utilizando kit comercial por qPCR
Dezoito amostras foram submetidas a genotipagem utilizando um kit comercial de qPCR (Kylt® PCV-2 Typing kit, SAN Group Biotech, Höltinghausen, Alemanha). Os resultados foram comparados com os obtidos no cPCR para avaliar a sensibilidade e especificidade. O kit comercial é um PCR multiplex em tempo real e diferencia o DNA viral de PCV2a, PCV2b e PCV2d. Os resultados positivos ou negativos de cada amostra, incluindo as coinfecções, foram submetidos ao teste Kappa de Cohen para avaliar a concordância entre os dois testes.
2.5 Comitê de ética
Todas as amostras de DNA já extraídas utilizadas neste estudo foram cedidas pelo laboratório do Centro de Diagnóstico de Saúde Animal (CEDISA) em Concórdia, Brasil de casos positivos de PCV2 e PCV3, não sendo necessário aprovação de comitê de ética.
3. Resultados
PCV2a, PCV2b, ou PCV2d foram detectados em 266 das 333 amostras testadas que foram submetidas a genotipagem, mas a detecção do genótipo não foi viável em 67 amostras, conforme mostrado na Tabela 2. De todas as 266 amostras genotipadas, 151 foram positivas para PCV2b (51,7%), 88 para PCV2d (30,1%) e 27 amostras apresentaram coinfecção para PCV2b e PCV2d (9,2%). Nenhuma amostra apresentou resultado positivo para PCV2a.
Tabela 2 Frequência e genótipos de PCV2 nas amostras analisadas.
O PCV2b foi detectado em amostras de cérebro (2/2), linfonodos (6/6), fezes (3/3) e útero (3/3) entre as amostras testadas. Por outro lado, as amostras de soro foram as mais frequentemente positivas para PCV2d, com 43 amostras positivas de um total de 81 amostras positivas (43/81). Portanto, pode-se observar o genótipo PCV2b em todos os tipos de materiais testados. Conforme mostrado na Tabela 2, as amostras de esfregaços retais apresentaram a maior percentagem de coinfecção entre os genótipos PCV2b e PCV2d.
Os oito estados brasileiros cujas amostras foram analisadas respondem por mais de 90% de toda a produção brasileira de carne suína(20). A maior parte das amostras foi recebida dos três estados do Sul (Paraná, Rio Grande do Sul e Santa Catarina), que representam a maior região produtora de suínos do país. Em relação à distribuição genotípica por estado, o PCV2b foi detectado com maior frequência em amostras do estado do Paraná (61,99%), enquanto o PCV2d foi mais frequente em amostras do estado de São Paulo. No Mato Grosso do Sul, onde foram observadas as maiores taxas de coinfecção entre os dois genótipos (PCV2b e PCV2d), e onde a genotipagem foi possível em 100% das amostras testadas (Figura 2).
Figura 2 Detecção do genótipo PCV2 por amostra por cada estado brasileiro, mostrando o genótipo mais frequente em cada amostra.
Em relação à fase de produção, a fase de terminação apresentou o maior número de amostras genotipadas, na qual o PCV2d foi o genótipo mais frequentemente detectado em 62 amostras, enquanto o PCV2b foi detectado em 41 amostras. Além disso, o PCV2d também foi mais frequente nas amostras de porcas. Na fase de creche, o genótipo mais frequente foi o PCV2b, presente em 51 amostras, enquanto o PCV2d foi detectado em 11 amostras. As maiores taxas de coinfecção de PCV2b e PCV2d foram encontradas nas amostras da fase de terminação, totalizando 20 amostras. As maiores taxas de PCV2b foram encontradas em amostras que não foram informadas em que fase de produção se encontravam, conforme mostra a Tabela 3.
Tabela 3 Detecção de genótipos de PCV2 por fase de produção, com os genótipos mais frequentes e o número total de amostras positivas.
3.1 Ocorrência de genótipos de PCV2 e/ou coinfecção por PCV3
Vinte e sete amostras apresentaram coinfecção entre os genótipos PCV2b e PCV2d, representando 9,2%. Entretanto, não foi encontrada nenhum outro tipo de coinfecção entre os genótipos de PCV2; a maioria das coinfecções foi encontrada em amostras obtidas de suabes, representando 66,67% das amostras positivas. Além disso, foram encontradas coinfecções com PCV3 em 26 amostras, tanto para PCV2 como para PCV3, o que representa 8,9%. As amostras de fetos apresentaram maior positividade para coinfecção entre PCV2 e PCV3. Além disso, foi observada maior presença de amostras do genótipo “b” nessas coinfecções, com 20 amostras positivas para PCV2b e 5 amostras positivas para PCV2d. Apenas uma amostra apresentou positividade para coinfecção entre PCV2b, PCV2d e PCV3 (dados não mostrados).
3.2 Avaliação da concordância entre os dois testes para genotipagem de PCV2
Foram escolhidas 18 amostras aleatoriamente, testadas com o kit comercial (Kylt® PCV2 Typing) e comparadas com cPCR. Os achados indicaram que a maioria das amostras apresentou coinfecção entre os genótipos (Tabela 4). Os resultados foram submetidos ao teste Kappa para avaliar a concordância entre os testes (Tabela 5).
Tabela 4 Comparação entre os dois testes de genotipagem para PCV2.
Tabela 5 Teste estatístico Cohen-Kappa para avaliar a concordância entre os dois testes.
A reação de cPCR foi mais sensível para algumas amostras, resultando na genotipagem bem-sucedida de quatro amostras. Apenas uma amostra (amostra 2) apresentou discrepância entre o kit comercial e o cPCR, sendo que o kit comercial identificou coinfecção (PCV2b e PCV2d), enquanto o teste de cPCR detectou apenas PCV2b. A análise dos dados entre os genótipos e os dois testes revelou resultados consistentes. O teste PCV2b e o PCV2 geral (cPCR e kit comercial) exibiram a mesma acurácia (77,8%). No teste PCV2d a acurácia foi maior (83,33%). O teste kappa apresentou intervalo de confiança maior que 0, indicando que ambos os testes conseguiram identificar os genótipos e mostrar concordância entre eles. Porém, a maior concordância foi encontrada na identificação do PCV2d, por apresentar acurácia e intervalo de confiança maior.
4. Discussão
Neste estudo, analisamos amostras clínicas de diagnóstico de PCVD que previamente apresentaram resultado positivo para PCV2 no laboratório CEDISA, laboratório de diagnóstico de sanidade animal, que recebe amostras para diagnóstico de doenças em suínos de todo o Brasil(22). Para complementar o diagnóstico das granjas vacinadas contra PCV2, porém com sinais de PCVD, no nosso estudo, 79,88% (266/333) das amostras de PCV2 foram genotipadas. Os resultados demonstraram que o PCV2b foi o genótipo predominante detectado em 151 das 266 amostras analisadas. Esta observação concorda com vários estudos a nível mundial, indicando que o PCV2b e o PCV2d são atualmente os genótipos mais prevalentes da infecção por PCV2 em suínos(11, 28). Além disso, nosso estudo demonstrou que os genótipos PCV2b e PCV2d foram encontrados em amostras de todos os estados brasileiros estudados, confirmando sua ampla ocorrência no país. Curiosamente, apesar da genotipagem de amostras positivas para PCV2 de casos clínicos, o PCV2a não foi detectado. Estudos anteriores demonstraram uma mudança nos genótipos predominantes na produção suína ao longo dos tempos, passando do genótipo PCV2a para PCV2b em 2002(29). Além disso, em 2012, ocorreu outra mudança de PCV2b para PCV2d(4, 13, 17). É importante lembrar que as vacinas baseadas em PCV2a são as mais utilizadas nos rebanhos suínos brasileiros (M. Donin, comunicação pessoal).
As coinfecções entre genótipos PCV2b e PCV2 estão se tornando mais frequentes(15, 17, 28, 30), e o mesmo foi observado em 27 amostras do nosso estudo. Também foi possível observar coinfecção com PCV3 em nosso estudo em 26 amostras. Novamente, este resultado está de acordo com estudos realizados em outros países, nos quais também foi possível identificar coinfecção entre PCV2 e PCV3(16, 17, 31). Contudo, nenhum dos estudos citados descreve qual genótipo do PCV2 foi predominantemente envolvido nas coinfecções com PCV3.
As amostras de linfonodos fornecem uma boa indicação de como está a infecção por PCV2 nas granjas de suínos(32). Em nosso estudo, a genotipagem foi possível para todas as amostras de linfonodos recebidas. Além do diagnóstico cuidadoso no momento da avaliação dos animais, a utilização de amostras de alta qualidade é de extrema importância. O PCV2b foi detectado em todos os tipos de amostras recebidas, indicando o seu papel na infecção sistêmica em animais. Por outro lado, o PCV2d foi detectado em amostras de pulmão, feto, intestino e soro. Um achado importante observado neste estudo foi a identificação de PCV2b em amostras de cérebro de suínos na terminação, porém as lesões cerebrais causadas pelo PCV2 podem ser ocasionais(4). No nosso estudo não foi possível correlacionar os sintomas clínicos apresentados pelos animais com o genótipo do PCV2 detectado.
O genótipo PCV2d foi o mais predominante na fase de terminação. Este resultado é consistente com um estudo realizado na Coreia do Sul, no qual foi encontrada uma maior prevalência de PCV2d em linfonodos de suínos no momento do abate(32). Por outro lado, na fase de creche, o PCV2b foi o genótipo mais comum. Até onde sabemos, este é o primeiro estudo a encontrar esses dados e buscar uma correlação entre genótipo e fase de produção. A terminação foi a fase em que foi possível obter e genotipar mais amostras previamente positivas para PCV2. Isto deve-se provavelmente à queda na imunidade da vacina e na imunidade materna, tornando os suínos mais susceptíveis às infecções por PCV2(32).
5. Conclusão
A genotipagem do PCV2 é importante para compreender o genótipo predominante em cada granja e discriminá-los, auxiliando no controle da doença e na monitoraria da eficácia da vacina. O PCV2b continua sendo o genótipo predominante nas granjas, juntamente com um aumento na detecção do genótipo PCV2d nas amostras avaliadas. É importante salientar a existência de coinfecções entre genótipos, incluindo coinfecção com PCV3. Com base nestes dados, deve ser dada maior atenção ao monitoramento da eficácia das vacinas e ao surgimento da doença clínica por circovírus. Assim, é importante estudar a evolução genética do PCV2 e avaliar a atualização das vacinas existentes no mercado, considerando a alta taxa de mutação apresentada pelo vírus. O controle da doença por circovírus deve combinar o uso adequado de vacinas com medidas de biossegurança e manejo sanitário, buscando melhorar a saúde dos suínos.
Declarações de conflito de interesse:
Os autores declaram não ter conflitos de interesse.
Disponibilidade de dados:
Os dados gerados nesta pesquisa estão publicados aqui e também podem ser solicitados aos autores.
Contribuições dos autores
Rovian Miotto: Metodologia, Curadoria dos dados, Escrita – Preparação do rascunho original, Validação, Análises dos dados. Caroline Pissetti: Metodologia, Curadoria dos dados, Validação, Recursos. Luiz Carlos Bordin: Investigação, Recursos. Janice Reis Ciacci Zanella: Supervisão, Conceitualização, Metodologia, Recursos, Escrita – Revisão e Edição, Administração do projeto, Aquisição de financiamento.
PUBLICADO ORIGINALMENTE NA REVISTA CIÊNCIA ANIMAL BRASILEIRA, EM JULHO DE 2024. DOI: 10.1590/1809-6891v25e-77826P
e-ISSN 1809-6891
Medicina Veterinária | Artigo científico
ACESSO DISPONÍVEL EM: https://revistas.ufg.br/vet/article/view/77826