Introdução
O mapeamento de QTLs (locos que controlam características quantitativas) através da análise de ligação envolve algumas etapas, dentre elas 1) o delineamento de uma população experimental (F2 ou retrocruzamento) através do cruzamento entre linhagens ou raças geneticamente divergentes, 2) a genotipagem, 3) a fenotipagem dos animais e, finalmente, 4) a implementação das análises genético-estatísticas.
Algumas abordagens têm sido empregadas para aumentar o poder de detecção e possibilitar que a identificação de QTLs na galinha doméstica seja facilitada, como por exemplo, a seleção de famílias e marcadores de DNA mais informativos (Zhu et al., 2001). Entretanto, a modelagem dos dados fenotípicos com base em modelos mistos não tem sido empregada rotineiramente no mapeamento de QTLs na galinha. Isto é devido ao fato de que o programa QTL Express (Seaton et al., 2002), que tem sido amplamente empregado no mapeamento de QTLs nesta espécie, considerar apenas efeitos fixos.
A modelagem fenotípica no mapeamento de QTLs foi proposta por Macgregor et al. (2005) em dados simulados e por Rodriguez-Zas et al. (2002) em gado leiteiro a partir de fenótipos longitudinais. Estes trabalhos demonstraram a importância de modelar apropriadamente os dados fenotípicos através da seleção de estruturas de co(variância) que pudessem explicar a maior parte da variância fenotípica.
Este trabalho relata o emprego de modelos mistos e a seleção da estrutura de co(variância) na modelagem dos dados fenotípicos da população experimental F2 delineada para mapeamento de QTLs associados a características de desempenho e carcaça da galinha.
Materiais & Métodos
Populações experimentais
A população TCTC foi desenvolvida através de cruzamentos entre a linhagem de corte (TT) e a linhagem de postura (CC) pela Embrapa Suínos e Aves, Concórdia, Santa Catarina, Brasil. Sete machos TT foram cruzados com sete fêmeas CC para a geração das aves F1 TC. Um total de sete machos e 21 fêmeas, correspondendo à proporção de um macho para três fêmeas de cada família da geração F1 foi selecionado para constituírem os parentais da geração F2, em que cada macho F1 foi acasalado com três fêmeas F1 não aparentadas. Cada fêmea F1 produziu aproximadamente 100 animais F2 por família de F1 em 17 incubações, totalizando cerca de 2.000 aves F2. Detalhes podem ser obtidos em Rosário et al. (2009).
A geração F2 foi anelada para controle de pedigree e avaliada para várias características de desempenho e de carcaça. As aves receberam rações formuladas à base de milho e farelo de soja, a fim de atender as necessidades nutricionais em cada período de criação. O manejo empregado foi da criação de frangos de corte. Ração e água foram fornecidas ad libitum.
As aves foram mantidas em galpões de criação até os 35 dias de idade, quando foram realizados a pesagem e alojamento em gaiolas individuais para a realização do teste de conversão alimentar dos 35 aos 41 dias. Aos 42 dias de idade realizou-se o abate para avaliação das características de carcaça.
Dados fenotípicos
Foram avaliadas as seguintes características fenotípicas nas aves F2: peso vivo (g) aos 35, 41 e 42 dias de idade; ganho de peso (g), consumo de ração (g), eficiência alimentar e conversão alimentar (g.g-1; valor absoluto) dos 35 aos 41 dias. Aos 42 dias de idade, as aves foram abatidas no Abatedouro Experimental de Suruvi, pertencente à Embrapa Suínos e Aves, Concórdia/SC, Brasil, de acordo com as normas sanitárias e de bem-estar animal para a obtenção dos pesos das partes (g) e dos órgãos (g): cabeça, pés, asas, pernas (coxas e sobrecoxas), peito (com pele e osso), dorso, gordura abdominal, fígado, coração, moela, pulmão e comprimento do intestino. O peso da carcaça eviscerada (g) (sem pés, cabeça, pescoço e gordura abdominal) foi obtido através da soma das partes. Parâmetros sanguíneos também foram avaliados: hematócrito (%), colesterol (mg.dL-1) e triglicerídeos (mg.dL-1).
Modelagem dos dados fenotípicos
Com a finalidade de selecionar o modelo linear que melhor se ajustava aos dados fenotípicos, considerou-se, em uma etapa inicial, o seguinte modelo misto no PROC MIXED do programa SAS® (SAS, 2007) para a análise:

O efeito de incubação foi considerado aleatório não devido ao processo em si, já que este é controlado (temperatura, umidade e tempo de rotação dos ovos), mas sim por considerar o efeito da idade da mãe entre as posturas. As fêmeas no decorrer de seu desenvolvimento apresentam uma curva de postura, na qual, pintos nascidos no início desta curva são mais leves do que aqueles nascidos no final da curva. Este fato é decorrente do tamanho dos ovos que no início da postura são menores que aqueles a partir do pico da curva (McLoughlin & Gous, 2000). Adicionalmente, como os animais F2 foram gerados ao longo de 17 incubações quinzenais (aproximadamente oito meses) e a mensuração dos fenótipos foram realizadas em tempos adjacentes, existe uma correlação maior entre os animais obtidos no início, no pico e no final da curva de postura. Este cenário se ajusta dentro do conceito de medidas repetidas no tempo. Uma possibilidade de se minimizar o efeito desta correlação intrínseca é modelar a estrutura da matriz de co(variância) para o efeito aleatório de incubação e do erro.
Para
Ik e e
ijkm foram consideradas as matrizes de co(variância)
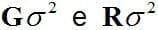
, respectivamente, as quais foram modeladas de acordo com sete estruturas: auto-regressiva de primeira ordem (AR1), auto-regressiva heterogênea de primeira ordem (ARH1), simetria composta (CS), simetria composta heterogênea (CSH), componentes de variância (VC), Toeplitz (TOEP) e Toeplitz heterogênea (TOEPH). A melhor estrutura foi selecionada com base no Critério de Informação de Akaike (AIC) (Akaike, 1974). Quanto menor o valor para este critério, mais apropriado é o modelo.
O peso vivo aos 35 dias foi utilizado como covariável para ganho de peso e para consumo de ração, conversão e eficiência alimentar dos 35 aos 41 dias. Para todas as características de carcaça, incluindo órgãos, foi empregado o peso vivo aos 42 dias como covariável.
Resultados & Discussão
Para a tomada de decisão sobre o modelo mais apropriado, foi assumido o peso vivo aos 42 dias como referência, pois esta característica apresentou alta correlação com as demais características (dados não mostrados).
Após a avaliação dos modelos com as combinações entre as estruturas da matriz de co(variância) testadas, foi selecionado aquele contendo apenas os efeitos de família, sexo e incubação, além da média e do erro. Para a matriz G, associada à incubação,a estrutura de co(variância) selecionada foi AR1 e para a matriz R, associada ao erro, foi VC, pois esta combinação foi a que apresentou menor valor para o AIC (Tabela 1). A estrutura AR1 é interessante, pois considera que a correlação entre cada incubação adjacente é reduzida ao longo do tempo (r1, r2, r3, ..., r17), ou seja, ela considera maior correlação entre duas quaisquer incubações adjacentes e menor correlacão entre duas quaisquer incubações distantes.
Tabela 1. Critérios de Informação de Akaike (AIC)
† para os efeitos aleatórios de incubação (
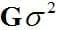
) e resíduo (

) para o peso vivo aos 42 dias de idade.
AR1: auto-regressiva de primeira ordem, ARH1: auto-regressiva heterogênea de primeira ordem, CS: simetria composta, CSH: simetria composta heterogênea, VC: componentes de variância, TOEP: Toeplitz e TOEPH: Toeplitz heterogênea.
† quanto menor o valor, melhor.
* matriz Hessian não positiva definida.
Devido às incubações terem sido realizadas quinzenalmente e as características mensuradas em tempos adjacentes, há uma maior correlação entre estas características e aquelas tomadas em tempos mais distantes. Dessa forma, os erros não são independentemente distribuídos, com média zero e variância
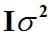
, violando um dos pressupostos da análise de variância. Por essa razão, utilizou-se o emprego do conceito de medidas repetidas no tempo e a modelagem da estrutura de co(variância), conforme Littell
et al. (1996), a fim de modelar apropriadamente os dados fenotípicos da população estudada.
Em análises de QTLs não é de nosso conhecimento trabalhos que modelaram a estrutura de co(variância) dos dados fenotípicos para o mapeamento de QTLs especificamente em galinhas. Já o mapeamento de QTLs envolvendo dados longitudinais foi feito por Macgregor et al. (2005) com simulação de dados e em dados reais de gado leiteiro por Rodriguez-Zas et al. (2002). Embora a estrutura de co(variância) AR1 não tenha sido avaliada em ambos os trabalhos, no presente estudo esta estrutura foi a escolhida, pois apresentou o menor valor de AIC, isto é, o modelo final apresentou o mais alto nível de informação com o menor número de parâmetros.
Através da modelagem fenotípica realizada neste trabalho, há a possibilidade de que tanto as posições quanto os efeitos dos QTLs que vierem a ser mapeados na população em estudo possam ser estimados de forma mais condizente com o delineamento experimental utilizado.
O próximo passo será investigar a magnitude das possíveis diferenças no mapeamento de QTL utilizando-se a incubação como efeito fixo e aleatório.
Conclusões
O emprego de modelos mistos e a seleção da estrutura de co(variância) na modelagem de dados fenotípicos oriundos da população F2 desenvolvida para mapeamento de QTLs demonstrou ser apropriado. Dessa forma, foi possível selecionar o modelo que apresenta o maior nível de informação com o menor número de parâmetros.
Agradecimentos
À FAPESP pela concessão de bolsas (processo número 10/50019-1) e ao CNPq e ao PRODETAB/EMBRAPA pelo auxílio financeiro.
Bibliografia
Akaike H. 1974. A new look at the statistical-model identification. IEEE Trans Autom Control 19: 716-723.
Littell RC, Milliken GA, Stroup WW, Wolfinger RD. 1996. SAS® system for mixed models. Cary: SAS Institute, 633p.
Macgregor S, Knott SA, White I, Visscher PM. 2005. Quantitative trait locus analysis of longitudinal quantitative trait data in complex pedigrees. Genetics 171:1365-1376.
McLoughlin L & Gous RM. 2000. Efecto del tamaño del huevo en el crecimiento pre y post natal de pollitos de engorde. Avicultura Professional (2):24-29.
Rodriguez-Zas SL, Southey BR, Heyen DW, Lewin HA. 2002. Detection of quantitative trait loci influencing dairy traits using a model for longitudinal data. J Dairy Sci. 85:2681-2691.
Rosário MF, Ledur MC, Moura ASAMT, Coutinho LL, Garcia AAF. 2009. Genotypic characterization of microsatellite markers in broiler and layer selected chicken lines and their reciprocal F1s. Sci Agric. 66:150-158.
SAS - Statistical Analyses System. 2007. SAS OnlineDoc®, version 9.13, Cary: SAS Institute. 1 CD-ROM.
Seaton G, Haley CS, Knott SA, Kearsey M, Visscher PM. 2002. QTL Express: mapping quantitative trait loci in simple and complex pedigrees. Bioinformatics 18:339-340.
Zhu JJ, Lillehoj HS, Cheng HH, Pollock D, Sadjadi M, Emara MG. 2001. Screening for highly heterozygous chickens in outbred commercial broiler lines to increase detection power for mapping quantitative trait loci. Poult Sci. 80:6-12.








