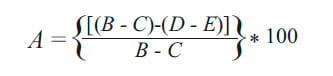1 Introdução
As aflatoxinas são metabólitos secundários de baixo peso molecular produzidas principalmente pelos fungos filamentosos1 Aspergillus flavus e A. parasiticus, assim como A. nominus, A. tamarii e A. pseudotamarii2. Elas apresentam efeitos tóxicos em humanos e animais, como efeitos carcinogênicos, mutagênicos, teratogênicos e imunosupressivos3, os quais são influenciados pela variação da espécie, sexo, idade, estado nutricional, além da dose e do período de exposição do organismo à toxina4. As aflatoxinas apresentam ocorrência mundial, principalmente nas áreas de clima tropical e subtropical, sendo as espécies de Aspergillus capazes de crescer em ampla variedade de substratos, como cereais, especiarias, oleaginosas e frutas secas, e sob diversas condições ambientais, principalmente em clima quente e úmido5,6.
São conhecidos, atualmente, 18 análogos de aflatoxina, sendo a aflatoxina B1 (AFB1) a mais prevalente e a mais tóxica7. Quando a AFB1 é ingerida por animais domésticos, entre eles o gado leiteiro, através do consumo de rações contaminadas, esta sofre biotransformação hepática convertendo-se em aflatoxina M1 (AFM1), que é excretada no leite, tecidos e fluidos biológicos desses animais8,9. Creppy10 relata que da totalidade de AFB1 ingerida através da ração, aproximadamente 0,3% a 6,2% é transformada em AFM1 no leite, dependendo de fatores nutricionais e fisiológicos, como regime de alimentação, taxa de ingestão, sanidade do animal, capacidade de biotransformação hepática, presença de infecções mamárias, período de lactação e produção de leite11.
A presença de AFM1 em leite e derivados pode trazer sérias consequências à saúde humana, já que o leite é o principal nutriente para o crescimento de crianças, cuja sensibilidade é notável e potencialmente maior que em adultos12. A ingestão diária média IDM de AFM1 estimada em países europeus é de 0,11 ng/kg de peso corpóreo (p.c.)/dia13. Oliveira et al.14 observaram a IDM de AFM1 igual a 3,7 ng/kg p.c./dia, para crianças da rede escolar pública da cidade de São Paulo. A legislação brasileira estabelece que os limites máximos de AFM1 admissíveis no leite fluido, leite em pó e queijos são 0,5 μg/L, 5,0 μg/kg e 2,5 μg/kg, respectivamente15. Diversos estudos demonstram a ocorrência frequente de AFM1 em leite e derivados no Brasil. No Estado de São Paulo, Gonçalez et al.16 pesquisaram a ocorrência de AFM1 em 43 amostras de leite pasteurizado, observando 11 (25,6%) delas com concentrações acima de 0,5 μg/L.
Portanto, faz-se necessária a busca por métodos de descontaminação que apresentem eficiência, especificidade e custo-benefício, que sejam ambientalmente corretos e possam ser produzidos em larga-escala17. A biodegradação de aflatoxinas utilizando microrganismos oferece uma alternativa atrativa para o controle ou eliminação dessas toxinas em alimentos e rações, mantendo sua qualidade e segurança18. Em revisão detalhada sobre o assunto, Bovo et al.19 explicam a capacidade das bactérias ácido-láticas (BAL) em remover eficientemente a AFB1 e AFM1 de meios contaminados. Diversos estudos têm sido realizados a fim de se conhecer a habilidade de remoção de aflatoxinas por esse tipo de bactéria20-23. Sendo assim, o objetivo do presente trabalho foi investigar a capacidade de algumas espécies de bactérias ácido-láticas em remover a AFM1 em tampão fosfato salino contaminado.
2 Material e Métodos
2.1 Cepas bacterianas e condições de cultivo
Nos testes de remoção de AFM1 em solução tampão fosfato salino (Phosphate Buffered Saline, PBS, pH 7,3) foram utilizadas 6 cepas de bactérias ácido-láticas gentilmente doadas pelo TECNOLAT – Centro de Pesquisa e Desenvolvimento de Laticínios do Instituto de Tecnologia de Alimentos (ITAL) de Campinas/SP (CTC204 – Lactococcus lactis ssp. cremoris, CTC483 – Enterococcus avium, PN16 – Lactobacillus paracasei, PN270 – Lactobacillus rhamnosus, TR260 – Lactobacillus plantarum, TR285 – Pediococcus pentosaceus). Todas as cepas bacterianas foram recebidas na forma liofilizada, reativadas em caldo MRS - Man, Rogosa & Sharpe (Acumedia® - Lansing, MI, USA) e conservadas a temperatura de -80 °C na presença de 20% de glicerol (Synth® - Diadema, SP, Brasil). As bactérias foram incubadas em caldo MRS a temperatura de 37 °C por 24 horas, sendo subcultivadas repetidas vezes até que alcançassem concentração elevada de células. A pureza das cepas foi examinada através de cultivo em ágar MRS (Acumedia® - Lansing, MI, USA) para verificação das colônias formadas e por coloração de Gram.
2.2 Estimativa da concentração bacteriana
Para realizar a estimativa da concentração de cada cepa bacteriana, foi construída uma curva relacionandose a densidade óptica do caldo de bactérias com 24 horas de crescimento a 37 °C, medida em espectrofotômetro Spectrumlab 22PC (Shanghai Lengguang Technology Co. Ltda – Shanghai, China) a 600 nm, e o logaritmo da concentração bacteriana obtido através da contagem de colônias por plaqueamento em profundidade24.
2.3 Ensaio de remoção de AFM1 em meio aquoso
Foi utilizado padrão de AFM1 (Supelco™ - Bellefonte, PA, USA), dissolvido em acetonitrila e calibrado espectrofotometricamente através da técnica preconizada por Scott25. A solução padrão foi diluída de maneira a obter uma solução de trabalho contendo 0,15μg AFM1/mL em PBS. Acetonitrila foi completamente evaporada por injeção direta de nitrogênio em banho a 45 °C. Todos os procedimentos de manipulação da AFM1 foram realizados em capela de exaustão com fluxo de segurança.
Cada cepa bacteriana foi incubada em caldo MRS a 37 °C por 24 horas e o crescimento interrompido por imersão em banho contendo água e gelo por 3 minutos. Um volume do caldo com a cultura de bactérias correspondendo a 5 x 109 UFC foi centrifugado (Microcentrífuga CT-14000, Cientec – Piracicaba, SP, Brasil) a 1.800g por 15 minutos. Os pellets formados foram lavados com água ultrapura (Milli-Q - Millipore® - Billerica, MA, USA) estéril, ressuspensos em 1,5 mL da solução de aflatoxina M1 em PBS e incubados a 37 °C por 15 minutos e 24 horas. Após a incubação, as células foram novamente centrifugadas a 1.800g por 15 minutos e o sobrenadante obtido foi analisado por cromatografia líquida de alta eficiência (CLAE). Para cada cepa bacteriana, um controle negativo (células bacterianas suspensas em PBS), um controle positivo (AFM1 em PBS) e um controle neutro (PBS) foram incubados e analisados. Todas as amostras foram analisadas em triplicata.
Os ensaios também foram realizados com as células bacterianas inviabilizadas. Para isso, após a incubação em caldo MRS a 37 °C por 24 horas, a cultura de bactérias foi mantida em banho a 100 °C por uma hora. A seguir, foi utilizado o mesmo procedimento descrito para as células viáveis.
2.4 Quantificação da AFM1 através de CLAE
Para a quantificação da AFM1, os sobrenadantes das culturas foram centrifugados a 1.800g por 15 minutos e diretamente injetados no sistema CLAE. Foi utilizado um equipamento constituído de detector de fluorescência RF- 10A XL (Shimadzu® - Tóquio, Japão), coluna Sinergy Fusion 4mm C18 4,6 X 150 mm (Phenomenex® - Torrance, CA, USA) e amostrador automático SIL-10AF (Shimadzu®). A fase móvel utilizada foi água:acetonitrila:metanol (60:20:20), fluxo de 1 mL/min, e detecção com comprimento de onda de excitação de 366 nm e emissão de 428 nm. O limite de detecção da técnica foi 0,01 ng/mL, considerando-se a concentração mínima de AFM1 necessária para gerar um pico cromatográfico 3 vezes maior que o desvio padrão da linha de base dos cromatogramas.
A quantificação da porcentagem de AFM1 adsorvida pela bactéria foi feita através da Equação 1, onde A representa a porcentagem de AFM1 adsorvida pela bactéria, B a área do pico cromatográfico do controle positivo, C a área do pico cromatográfico do controle neutro, D a área do pico cromatográfico da amostra e E a área do pico cromatográfico do controle negativo.
2.5 Análise estatística
Os resultados obtidos foram submetidos à análise de variância, de acordo com os procedimentos estabelecidos no General Linear Model do SAS26, para a verificação de diferenças estatisticamente significativas entre as cepas bacterianas e as condições de experimento. Para comparação entre as médias, quando aplicável, empregou-se o teste de Tukey, adotando-se nível de rejeição de 5%27.
3 Resultados e Discussão
As curvas de concentração bacteriana foram construídas através da correlação entre as medidas de absorbância obtidas e do plaqueamento em profundidade de cada bactéria. A partir dos dados obtidos, geraram-se equações potenciais, que foram utilizadas para o cálculo da concentração bacteriana no meio e, consequentemente, para o cálculo do volume de meio com o cultivo de bactérias utilizado para alcançar uma concentração de células de aproximadamente 5 x 109 UFC. As equações adequaram-se perfeitamente aos dados, já que o valor dos coeficientes de determinação (R2) de cada uma delas variou entre 0,997 e 0,999.
As porcentagens de remoção de AFM1 pelas BAL foram obtidas comparando-se os cromatogramas gerados pelo sistema CLAE, tanto para as células bacterianas viáveis quanto para as inviáveis a 37 °C nos tempos de contato de 15 minutos e 24 horas. O tempo de retenção da AFM1 nas condições de análise especificadas foi de 6,1 minutos. Os resultados encontrados para os ensaios de remoção de AFM1 em PBS estão descritos na tabela 1.
Comparando-se os resultados encontrados para os diferentes tratamentos aplicados, observa-se que, de maneira geral, as células inviáveis apresentaram as maiores porcentagens de remoção da AFM1, tanto no tempo de 15 minutos quanto no tempo de 24 horas, exceto para Lb. Paracasei, que não apresentou diferença significativa entre os tratamentos (p>0,05). Quando analisadas as diferenças de remoção de AFM1 entre as bactérias estudadas, observouse que, para as células viáveis no tempo de 15 minutos não houve diferenças significativas entre as bactérias (p>0,05). Entretanto, para as células inviáveis no mesmo tempo de remoção (15 minutos), Lb. plantarum apresentou a maior taxa de remoção, embora não tenha diferido significativamente de Lb. paracasei e Lb. rhamnosus. Já no tempo de contato de 24 horas, as células viáveis de P. pentosaceus mostraram-se as mais eficazes em remover a AFM1 do meio, enquanto células inviáveis de E. avium apresentaram o maior valor de remoção da toxina, embora este não foi significativamente diferente dos valores de Lc. cremoris, Lb. rhamnosus e Lb. plantarum.
Tabela 1: Porcentagem de remoção1 de AFM1 em solução tampão fosfato
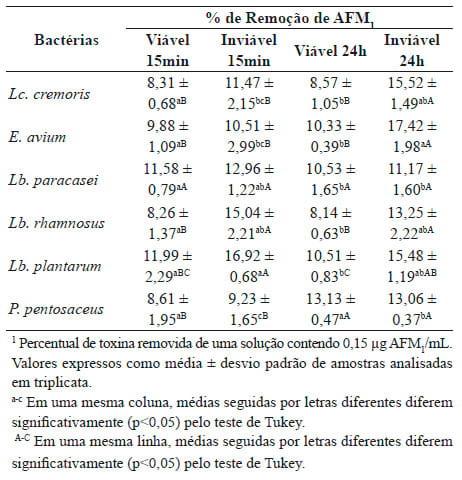
Elgerbi et al.28 analisaram a capacidade de remoção de AFM1 por células viáveis de 12 cepas de BAL em PBS a 37 °C com as contagens bacterianas variando entre 1,3 x 108 e 5,0 x 109 UFC/mL. Após 24 horas de contato, os resultados variaram entre 0% e 14,6%, sendo Lactococcus lactis ssp. cremoris (NCFB 1986) responsável pelo maior resultado, enquanto que Lactobacillus rhamnosus (ATCC 7469) e Lactobacillus plantarum (DSM 12028) removeram 0,6% e 0,0%, respectivamente. Quando esses valores foram comparados com os observados na tabela 1 para as mesmas espécies, embora para cepas diferentes, percebeu-se que Lc. cremoris teve resultado inferior, enquanto que as outras duas bactérias tiveram resultados mais elevados (8,57%, 8,14% e 10,51%, respectivamente). Assim, observa-se que dentro de um dado gênero e até mesmo de determinada espécie, nem todas as cepas são equivalentes em termos de remoção da toxina, ao contrário, a capacidade de remoção de aflatoxina é característica apenas de linhagens específicas, com sua eficácia variando acentuadamente29.
Pierides et al.21 analisaram a habilidade de células viáveis e tratadas termicamente de 8 cepas de BAL em remover a AFM1 em tampão fosfato a 37 °C após 16 horas de contato e obtiveram resultados variando de 18,1 a 53,8% para as células viáveis e de 25,5 a 61,5% para as inviáveis. Os pesquisadores observaram que Lactobacillus rhamnosus (cepas GG e LC705) foram as bactérias que apresentaram as melhores taxas de remoção, tanto para as células viáveis (50,7% e 46,3%, respectivamente) quanto para as células inviáveis (57,8% e 51,6%, respectivamente). El-Nezami et al.20, trabalhando com as mesmas bactérias, obtiveram resultados de remoção de AFB1 de 77,0% e 65,0%, respectivamente. Segundo Pierides et al.21, a remoção da AFM1 foi menos efetiva possivelmente devido à presença de um grupo –OH adicional na molécula, o que resulta em aumento da polaridade da mesma, tornando-a mais hidrofílica e aumentando sua tendência em ficar retida em soluções aquosas.
Kabak e Var23 examinaram a capacidade de 4 cepas de Lactobacillus e 2 cepas de Bifidobacterium em remover a AFM1 em solução tampão e observaram que as células viáveis (108 UFC/mL) foram capazes de remover de 10,22% a 26,65% da AFM1 presente na solução, dependendo do nível de contaminação e do período de incubação, enquanto que as células inviáveis removeram de 14,04% a 28,97% da toxina. Os pesquisadores concluíram que o processo de remoção foi rápido, não havendo diferenças significativas entre os tempos de contato de 0, 4 e 24 horas. Já Elgerbi et al.28 observaram que cepas de Lactobacillus, Lactococcus e Bifidobacterium apresentaram porcentagens de remoção variando entre 0% e 14,6% no tempo de 24 horas, e entre 4,5% a 73,1% no tempo de 96 horas.
Observou-se que a viabilidade bacteriana não foi prérequisito para a remoção de AFM1 pelas BAL e que alguns tratamentos de inativação, sejam eles físicos, químicos ou enzimáticos, são capazes de aumentar a habilidade das BAL em unir-se à aflatoxina do meio. Haskard et al.30 estudaram a capacidade de L. rhamnosus GG em unir-se à AFB1, observando pouca diferença entre a remoção de aflatoxina pelas células tratadas termicamente e com ácido (85% e 91%, respectivamente), em relação às células viáveis da bactéria (86%). Os autores também observaram que a adição de uréia ao meio, um agente antihidrofóbico, diminuiu significativamente a remoção da toxina pelas células inviáveis, de 85 a 91% para 50 a 60%, mostrando que interações hidrofóbicas possuem papel relevante. Além disso, a adição de diferentes concentrações de NaCl e CaCl2 (de 0,01 a 1M) e variação de pH de 2,5 a 8,5 praticamente não apresentaram efeitos sobre a remoção da AFB1 pela bactéria, sugerindo que ligações de hidrogênio e interações eletrostáticas não são importantes.
Azab et al.31, avaliando a influência de tratamentos de inativação sobre a capacidade de 4 tipos de Lactobacillus em remover a AFB1, observaram que os tratamentos ácido (58,6% a 87,0%) e térmico (33,5% a 71,9%) aumentaram a capacidade de remoção quando comparados à solução tampão (16,3% a 56,6%). Já os tratamentos, alcalino (8,3% a 27,4%) e com etanol (15,9% a 46,5%) diminuíram a quantidade de aflatoxina removida.
Apesar do mecanismo de ação das bactérias sobre as aflatoxinas não ser completamente esclarecido, supõe-se que ocorra ligação de natureza física aos componentes da parede celular bacteriana, principalmente aos polissacarídeos e aos peptidoglicanos, já que tanto células viáveis quanto inviáveis foram capazes de unir-se à micotoxina, descartando-se a união através de ligações covalentes ou degradação pelo metabolismo da bactéria32.
Os tratamentos de inviabilização causam perturbações à parede celular bacteriana, permitindo que a aflatoxina se vincule aos componentes da membrana celular plasmática que estavam indisponíveis quando esta estava intacta. O tratamento com ácidos rompe ligações glicosídicas em polissacarídeos e proteínas, transformando-os em componentes menores e quebrando a estrutura do peptidoglicano, reduzindo ligações cruzadas e aumentando o tamanho dos poros. No entanto, o tratamento térmico causa a desnaturação de proteínas, aumentando a natureza hidrofóbica de sua superfície e formando produtos da reação de Maillard22. Hernandez- Mendoza et al.33 explicam que a integridade da parede celular bacteriana é importante no processo de remoção de aflatoxina por células viáveis e inviáveis. Em seu estudo com AFB1, os autores puderam verificar que tanto a parede celular bacteriana quanto seus fragmentos purificados foram capazes de remover a aflatoxina do meio, entretanto, quando ocorreu a perda ou destruição da parede celular (total ou parcial) em resposta a tratamentos enzimáticos, uma diminuição significativa da capacidade de remoção foi observada.
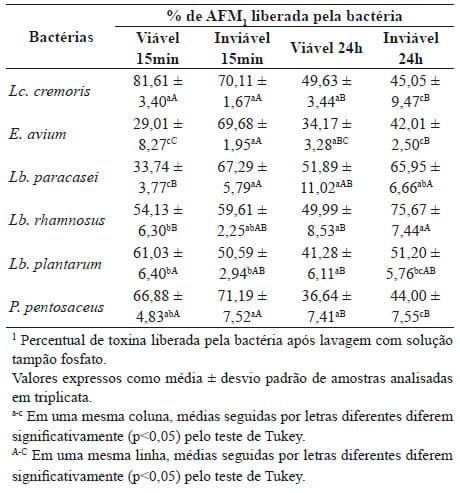
Observa-se na tabela 2 que as quantidades de AFM1 liberadas de volta para a solução tampão após lavagem da bactéria variaram de 29,01% a 81,61%. As células viáveis de Lc. cremoris com tempo de contato de 15 minutos foram as que mais liberaram AFM1 de volta para o meio, embora não tenham diferido significativamente de P. pentosaceus e do resultado apresentando por suas células inviáveis no mesmo tempo de contato. A bactéria que menos liberou a AFM1 para a solução foi E. avium, no tratamento com células viáveis e tempo de 15 minutos. Entretanto, esse valor não apresentou diferenças significativas em relação às suas células viáveis no tempo de 24 horas e ao valor encontrado para as células viáveis de Lb. paracasei no tempo de 15 minutos.
Elgerbi et al.28 também observaram grande liberação da AFM1 após a primeira lavagem dos pellets bacterianos com PBS (72,7% a 98,7%), sendo que após a terceira lavagem, praticamente toda a AFM1 adsorvida havia sido liberada de volta para o meio pelas bactérias analisadas (92% a 100%). Contrariamente, Kabak e Var23 observaram que apenas pequenas quantidades de AFM1 voltaram para a solução tampão (5,62% a 8,54%) após uma lavagem.
Tabela 2: Porcentagem1 de AFM1 liberada pelas cepas bacterianas após lavagem com PBS
Observa-se grande variação em estudos realizados para avaliar o efeito da lavagem de pellets bacterianos sobre a liberação de AFB1. Enquanto Haskard et al.22 observaram que L. rhamnosus GG, L. rhamnosus LC-705 e Lactobacillus casei Shirota liberaram 3,7%, 3,0% e 2,4%, respectivamente, da AFB1 de volta para a solução; Peltonem et al.34 encontraram valores de liberação da AFB1 na primeira lavagem de 48,6%, 30,7% e 26,5% para Lactobacillus amylovorus (cepas CSCC 5160 e CSCC 5197) e L. rhamnosus Lc 1/3, respectivamente, e após 5 lavagens, 94,4%, 82,6% e 67,8%, respectivamente.
Portanto, a capacidade de retenção de aflatoxina por BAL é instável e quanto maior o número de lavagens, maior a quantidade de toxina liberada para a solução, mostrando que a ligação é não-covalente fraca e que ocorre uma associação aos sítios hidrofóbicos na superfície bacteriana30. Embora a estrutura da parede celular dos diferentes tipos de BAL seja parecida, existem pequenas diferenças nos sítios de ligação presentes em cada cepa que faz com que exista essa variação na adsorção e na liberação da aflatoxina. Hernandez- Mendoza et al.33 explicam que a menor liberação da toxina para o meio após as lavagens pode ser atribuída às interações entre as moléculas de aflatoxina retidas na parede celular de uma bactéria com as moléculas retidas na parede celular da bactéria adjacente, formando uma espécie de matriz reticulada que impede a liberação da aflatoxina.
4 Conclusão
Os resultados encontrados demonstram que as cepas de bactérias ácido-láticas estudadas não apresentaram elevada eficiência de remoção da AFM1 em solução PBS. Contudo, pode-se concluir que a viabilidade bacteriana não é um requisito para que ocorra a remoção da toxina e que o processo de adsorção ocorre rapidamente, já que as células inviáveis foram mais eficazes e não se observou diferença entre os tempos analisados. Sendo assim, faz-se necessária a pesquisa de novas cepas bacterianas, e de um maior entendimento dos mecanismos de ação destas sobre as aflatoxinas para que se possam identificar aquelas que são eficazes na remoção das micotoxinas e, assim, ajudem a reduzir a exposição humana e animal a esses contaminantes.
Referências
1. Gonçalez E, Pinto MM, Felicio JD. Análise de micotoxinas no Instituto Biológico de 1989 a 1999. Biológico 2001;63(1/2):15-9.
2. Alberts JF, Engelbrecht Y, Steyn PS, Holzapfel WH, van Zyl WH. Biological degradation of aflatoxin B1 by Rhodococcus erythropolis cultures. Int J Food Microbiol 2006;109:121-6.
3. Hernandez-Mendoza A, Garcia HS, Steele JL. Screening of Lactobacillus casei strains for their ability to bind a?atoxin B1. Food Chem Toxicol 2009;47:1064-8.
4. Mishra HN, Das C. A Review on biological control and metabolism of aflatoxin. Crit Rev Food Sci Nutr 2003;43(3):245-64.
5. Park DL, Liang B. Perspectives on aflatoxin control for human food and animal feed. Trends Food Sci Technol 1993;41:334-342.
6. Bhat R, Rai RV, Karim AA. Mycotoxins in food and feed: present status and future concerns. Comp Rev Food Sci Food Saf 2010;9:57-81.
7. Oliveira CAF, Germano PML. Aflatoxinas: conceitos sobre mecanismos de toxicidade e seu envolvimento na etiologia do câncer hepático celular. Rev Saúde Pública 1997;31(4):417-4.
8. Oatley JT, Rarick MD, Ji GE, Linz JE. Binding of a?atoxin B1 to bi?dobacteria in vitro. J Food Protection 2000;63(8):1133–36.
9. Murphy PA, Hendrich S, Landgren C, Bryant CM. Food mycotoxins: an update. J Food Sci 2006;71(5):51-65.
10. Creppy EE. Update of survey, regulation and toxic effects of mycotoxins in Europe. Toxicol Letters 2002;127:19-28.
11. Fink-Gremmels J. Mycotoxins in cattle feeds and carry-over to dairy milk: a review. Food Addit Contam 2008;25(2):172- 80.
12. Prandini A, Tansini G, Sigolo S, Filippi L, Laporta M, Piva G. On the occurrence of aflatoxin M1 in milk and dairy products. Food Chem Toxicol 2009;47:984-91.
13. World Health Organization. Evaluation of certain mycotoxins. Geneva: WHO; 2001.
14. Oliveira CAF, Germano PM, Bird C, Pinto CA. Immunochemical assessment of aflatoxin M1 in milk powder consumed by infants in São Paulo, Brazil. Food Addit Contam 1997;14:7-10.
15. Brasil. Ministério da Saúde. Resolução RDC no7, de 18 de fevereiro de 2011 da ANVISA. Dispõe sobre limites máximos tolerados (LMT) para micotoxinas em alimentos. Diário Oficial da União 22 fev 2011.
16. Gonçalez E, Felicio JD, Pinto MM, Rossi MH, Nogueira JHC, Manginelli S. Ocorrência de Aflatoxina M1 em leite comercializado em alguns municípios do estado de São Paulo. Arq Inst Biol 2005;72:435-8.
17. Wu Q, Jezkova A, Yuan Z, Pavlikova L, Dohnal V, Kuca K. Biological degradation of aflatoxins. Drug Metab Rev 2009;41(1):1-7.
18. Alberts JF, Gelderblomb WCA, Botha A, van Zyl WH. Degradation of a?atoxin B1 by fungal laccase enzymes. Int J Food Microbiol 2009;135:47-52.
19. Bovo F, Corassin CH, Oliveira CAF. Descontaminação de aflatoxinas em alimentos por bactérias ácido-láticas. UNOPAR Cient Ciênc Biol Saúde 2010;12(2):15-21.
20. El-Nezami H, Kankaanpää P, Salminen S, Ahokas J. Ability of dairy strains of lactic acid bacteria to bind a common food carcinogen, aflatoxin B1. Food Chem Toxicol 1998;36:321-326.
21. Pierides M, El-Nezami H, Peltonen K, Salminen S, Ahokas J. Ability of dairy strains of lactic acid bacteria to bind aflatoxin M1 in a food model. J Food Prot 2000;63(5):645-50.
22. Haskard CA, El-Nezami HS, Kankannpää PE, Salminen S, Ahokas JT. Surface binding of aflatoxin B1 by lactic acid bacteria. Appl Environ Microbiol 2001;67(7):3086-91.
23. Kabak B, Var I. Factors affecting the removal of aflatoxin M1 from food model by Lactobacillus and Bifidobacterium strains. J Environ Sci Health Part B. 2008;43:617-24.
24. American Public Health Association. Standard methods for the examination of dairy products. Washington: APHA; 2004.
25. Scott PM. Natural poisons. In: Helrich, K. Official methods of analysis of the Association of Official Analytical Chemists. Arlington: AOAC; 1990.
26. SAS Institute. SAS User's Guide: statistics. Cary, NC: SAS; 1992.
27. Gacula JR, Singh J. Statistical methods in food and consumer research. Orlando: Academic; 1984.
28. Elgerbi AM, Aidoo KE, Candlish AAG, Williams AG. Effects of lactic acid bacteria and bifidobacteria on levels of aflatoxin M1 in milk and phosphate buffer. Milchwissenschaft 2006;61(2):197-9.
29. El-Nezami H, Mykkänen H, Haskard C, Salminen S, Salminen E. Lactic acid bacteria as a tool for enhancing food safety by removal of dietary toxins. In: Salminen S, von Wright A, Ouwehand A. Lactic acid bacteria: microbiological and functional aspects. Nova Iorque: Marcel Dekker; 2004.
30. Haskard C, Binnion C, Ahokas J. Factors affecting the sequestration of a?atoxin by Lactobacillus rhamnosus strain GG. Chem Biol Int 2000;128:39-49.
31. Azab RM, Tawakkol WM, Hamad ARM, Abou-Elmagd MK, El-Agrab HM, Refai MK. Detection and estimation of aflatoxin B1 in feeds and its biodegradation by bacteria and fungi. Egyp J Natl Toxins 2005;2:39-56.
32. Lahtinen SJ, Haskard CA, Ouwehand AC, Salminen SJ, Ahokas JT. Binding of a?atoxin B1 to cell wall components of Lactobacillus rhamnosus strain GG. Food Addit Contam 2004;21(2):158-64.
33. Hernandez-Mendoza A, Guzman-de-Peña D, Garcia HS. Key role of teichoic acids on aflatoxin B1 binding by probiotic bacteria. J Appl Microbiol 2009;107:395-403.
34. Peltonen K, El-Nezami, H, Haskard C, Ahokas J, Salminen S. Aflatoxin B1 binding by dairy strains of lactic acid bacteria and bifidobacteria. J Dairy Sci 2001;84:2152-6.